
近日,华南师范大学环境学院应光国教授团队史文俊副研究员等人在《Journal of Hazardous Materials》上发表了题为“Predicting estrogen receptor agonists from plastic additives across various aquatic-related species using machine learning and AlphaFold2”的论文(https://doi.org/10.1016/j.jhazmat.2025.138629)。该研究通过机器学习和AlphaFold2高通量预测多个水生相关物种的雌激素受体激动剂。
全文速览
由于缺乏水生生物相关的公共数据库极大地限制了高通量预测核受体介导激素效应。在本研究中,我们结合机器学习和AlphaFold2开发了筛选多物种雌激素受体(ER)激动剂的新策略。首先,使用机器学习(ML)模型对ERα激动剂进行筛选,共筛选42种塑添加剂为潜在的ER激动剂。然后,利用AlphaFold2构建斑马鱼(Danio rerio, Dr)、青鳉鱼(Oryzias melastigma, Om)、海豚(Delphinus delphis, Dd)、抹香鲸(Physeter catodon, Pc)、贻贝(Mytilus edulis, Me)、热带爪蟾(Xenopus tropicalis, Xt)、朱鹮(Nipponia nippon, Nn)和帝企鹅(Aptenodytes forsteri, Af)的ERα结构。结果显示,除了Me之外,大多数物种有两个共同的氨基酸残基与ERα活性密切相关:精氨酸85和谷氨酸44(LBD序列对齐后的序列编号,对应人类ER受体中精氨酸394和谷氨酸353)。然而,水生相关物种还显示出另外三个额外的关键残基:甘氨酸212、亮氨酸216和苯丙氨酸95(LBD序列对齐后的序列编号)。高通量分子对接结果显示Dr、Om、Dd、Pc、Me、Xt、Nn和Af对接能量< -9 kcal/mol的塑料添加剂数量分别为4、8、4、12、10、13、7和9。所有物种中,雌酮的对接能量均< -9 kcal/mol,而双酚P在不同物种中的对接能量差异较大。以上结果表明,ML与AlphaFold的联合应用可高通量预测新污染物与多种水生相关物种中核受体的亲和力。
引言
新污染物(如塑料添加剂)种类和消耗量的剧增,导致其在环境中的检测频率和检出频率显著上升。不同的物种暴露于多种多样的污染物之下,这对它们的健康和整体生存状况构成了重大威胁。例如,北极捕食者如海豹、鲸鱼和北极熊正面临令人担忧的高浓度污染物暴露。传统的实验方法,如体外和体内实验,往往需要高成本的投入、面临通量低,还涉及动物实验伦理等问题。此外,包括极地生物、海洋生物和稀有物种在内的物种并不适合进行大规模的动物实验。美国环保署已宣布计划到2025年将哺乳动物测试减少30%。而通过计算机模拟方法评估毒性得到了广泛应用。基于污染物结构特性的筛选方法因其能够快速且经济高效地预测毒理终点,尤其是通过机器学习(ML)技术,受到了广泛关注。
前期研究主要预测污染物对某一种生物的毒性。然而,不同物种之间存在的显著生态差异会导致在接触不同物质时表现出显著差异的毒性效应。例如, 6PPD-Q对虹鳟鱼(Oncorhynchus mykiss)和红点鲑鱼(Salvelinus fontinalis)表现出急性毒性,其24小时半致死浓度(LC50)分别为1.96 μg/L和0.59 μg/L。相比之下,它对斑马鱼和青鳉未表现出显著的急性致死毒性。同样,氧化锌(ZnO)纳米颗粒对不同无脊椎动物和鱼类物种的急性毒性差异较大,范围从40 μg/L到58 mg/L。因此,评估不同物种对新兴污染物的敏感性对于更广泛理解这些新污染物对物种的易感性至关重要。分子对接技术是评估新兴污染物与蛋白质相互作用的生态毒性的重要方法之一。由DeepMind开发的AlphaFold方法表现出卓越的性能,其在CASP14测试中取得的成功被广泛认可为解决蛋白质结构预测问题的方案。凭借其高度的准确性和快速的预测速度,AlphaFold构建了大规模结构预测的综合数据库,已被广泛应用于药物筛选和人类疾病治疗,但其在研究新污染物跨物种生态毒性中的应用仍然较少。而在生态毒理学中,跨物种评估新污染物毒性对理解其对生态系统的影响至关重要。
雌激素受体是污染物常见的结合靶点,许多新污染物通过与ER结合影响生物正常的转录和信号通路。由于缺乏针对生态物种的公共数据库以及物种预测的多样性有限,本研究提出了一种将机器学习模型与分子建模相结合的新策略,用于快速筛选不同物种的ER激动剂。在本研究中,我们首先构建了ML模型以识别塑料添加剂中的ER激动剂。随后,利用AlphaFold2,我们构建了包括鱼类、两栖动物、水鸟、海洋生物和南极生物在内的多种物种的ER结构。通过分子对接,我们分析了可疑ER激动剂与不同物种ER的结合亲和力和相互作用模式。
图文导读
1. 预测流程
ER激动剂的跨物种筛选流程在图1所示,包括以下步骤:1) 数据集的收集和清洗。2) 分子指纹和分子描述符生成与选择。3) 传统机器学习、深度森林和人工神经网络模型的训练与测试。4) 使用最佳模型预测ER激动剂。5) 多物种ER序列比对。6) 利用AlphaFold2构建各种物种的ER蛋白模型。7) 分子对接识别与ER激动剂作用相关的关键氨基酸残基。8) 根据分子对接结果筛选高亲和力化合物。

图1. 通过机器学习和分子模型预测不同物种雌激素受体激动剂流程
2. 不同机器学习方法构建模型的性能
本文使用6种机器学习方法,15种输入特征共构建了90个机器学习模型(图2A;SI_Text表S5)。在测试数据集上,平衡准确率(ACC)、灵敏度(sensitivity)、特异性(specificity)、F1分数和AUC的范围分别为0.76到0.98、0.57到0.96、0.85到1.00、0.49到0.98和0.85到1.00(图2A;SI_Text表S5)。其中,使用深度森林(Deep Forest, DF)算法有3个模型表现出卓越的性能(所有指标均超过0.9)。使用随机森林(RF)、逻辑回归(LR)、支持向量机(SVM)、K近邻(KNN)或人工神经网络(ANN)构建的模型均未达到这一性能水平(SI_Text表S5)。
但是,EstateFingerprint 和 AtomPairs2DFingerprint 经常导致模型性能不佳且不平衡。相比之下,使用 RDKit.Chem.Descriptors、MorganFingerprint、KlekotaRothFingerprintCount 和 KlekotaRothFingerprint 作为输入特征,在各种特征集上均表现出更好的性能(图 2B,SI_Text 表 S5)。例如,当使用 EstateFingerprint 作为输入特征时,DF、RF、LR、SVM、KNN 和 ANN 的 F1 分数分别为 0.55、0.64、0.52、0.56、0.49 和 0.51。而当使用 MorganFingerprint 作为输入特征时,DF、RF、LR、SVM、KNN 和 ANN 的 F1 分数显著提升,分别为 0.95、0.85、0.79、0.90、0.71 和 0.83。
总之,使用 RDKit.Chem.Descriptors 和 MorganFingerprint 作为输入特征在 DF 模型性能中排名前两位,它们的ACC = 0.98,灵敏度(sensitivity)= 0.96,特异性(specificity)> 0.99,F1 分数 > 0.95 和 AUC = 1.00(图 2B,SI_Text 表 S5)。
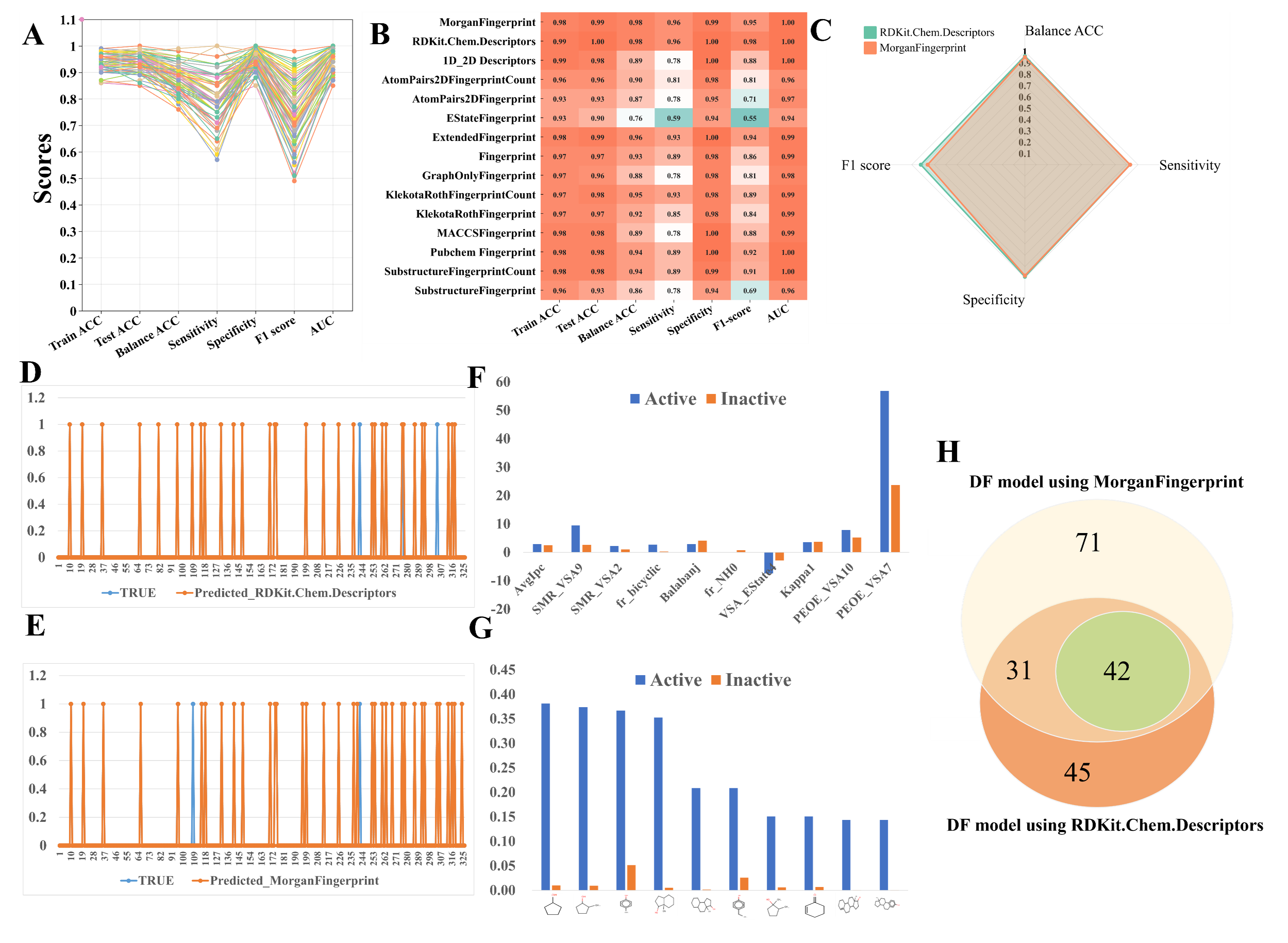
图2. 机器学习模型和ER激动剂筛选。(A) 机器学习模型的性能分布。(B) DF模型性能。(C) 使用RDKit.Chem.Descriptors和MorganFingerprint 构建的DF模型在验证集上性能。(D, E) 验证集中,使用RDKit.Chem.Descriptors (D)和MorganFingerprint (E)的DF模型预测输出值与真实值的比较。(F) 活性和非活性数据集中前10个重要特征的平均值。(G) 使用MoSS子结构分析。(H) 根据两种最优DF模型预测塑料添加剂列表中ERα激动剂的数量。ACC: 准确率;AUC: 曲线下面积;深度森林: DF;随机森林: RF;K近邻分类器: KNN;逻辑回归: LR;支持向量机: SVM;人工神经网络: ANN。
3. 重要特征和子结构
通过分子描述符特征重要性分析显示,排名前十的分子描述符分别是Avglpc、SMR_VSA9、SMR_VSA2、fr_bicyclic、Balabanj、f_NH0、VSA_EState4、Kappa1、PEOE_VSA10和PEOE_VSA7,其相对重要性数值均大于0.04(见SI_Text图S3)。同时,前十重要特征的平均值在活性化合物和非活性化合物之间表现出显著差异,反映了这些特征对ER活性的影响(图2F)。
MoSS功能节点分析识别出在活性和非活性化合物之间34种不同的子结构片段(见SI_Text表S6)。羟基脂环烃基团占活性样本中物质的38%,但在非活性样本中仅占1%。活性样本中酚基、脂环烃和酮基的比例分别为37%、37%和35%(图2G,SI_Text表S6)。
4. 筛选PLASTICMAP列表中可疑的ERα激动剂
使用基于RDKit.Chem.Descriptors和MorganFingerprint的DF模型,共识别出73个化合物被两个模型同时预测为ERα激动剂(SI_Dataset 表S7)。在去除与训练数据集重叠的化合物后,最终获得了42个可疑的ERα激动剂(图2H,SI_Dataset 表S8)。这42个可疑的激动剂与训练数据集的最大相似性范围为0.21至0.95。选取三种预测的ER激动剂进行暴露实验,结果显示在 14 dpf 时,单一暴露于 BPA、PRO 和 TRI 显著增加了与 ER 通路相关基因的转录表达水平,包括 cyp11a、cyp19a1a、esr1、foxl2 和 vtg1(见SI_Text 图 S4)。暴露于可疑 ER 激动剂与 雌激素受体激动剂FUL 的二元混合物表现出拮抗效应。例如,cyp11a、cyp19a1a、esr1、foxl2 和 vtg1 的转录均被FUL 抑制(见表 1)。
5. 不同物种的ERα结构
多物种ER序列比对与系统发育分析:全长氨基酸序列比对显示出显著的物种间多样性(SI_Text 图 S5-7)。Hs ERα与其他物种之间的序列相似性范围为32.33%至98.47%。相比之下,Me ERα与其他物种的相似性显著较低,范围为30.37%至34.50%(SI_Text 表 S7)。
在配体结合域(LBD)氨基酸序列比对中观察到类似的现象(SI_Text 表 S8)。所有9个物种的LBD氨基酸序列比对结果见SI_Text 图 S7。由于物种较多,为了方便比较氨基酸的保守性以及便于读者阅读,本论文中的氨基酸序号均是基于LBD序列对齐后序列编号。
总体上脊椎动物物种聚集在一个系统发育分支上(例如Hs、Dd和Pc;Dr和Om;Nn和Af),而无脊椎动物Me则表现出明显不同的进化轨迹(图3A)。
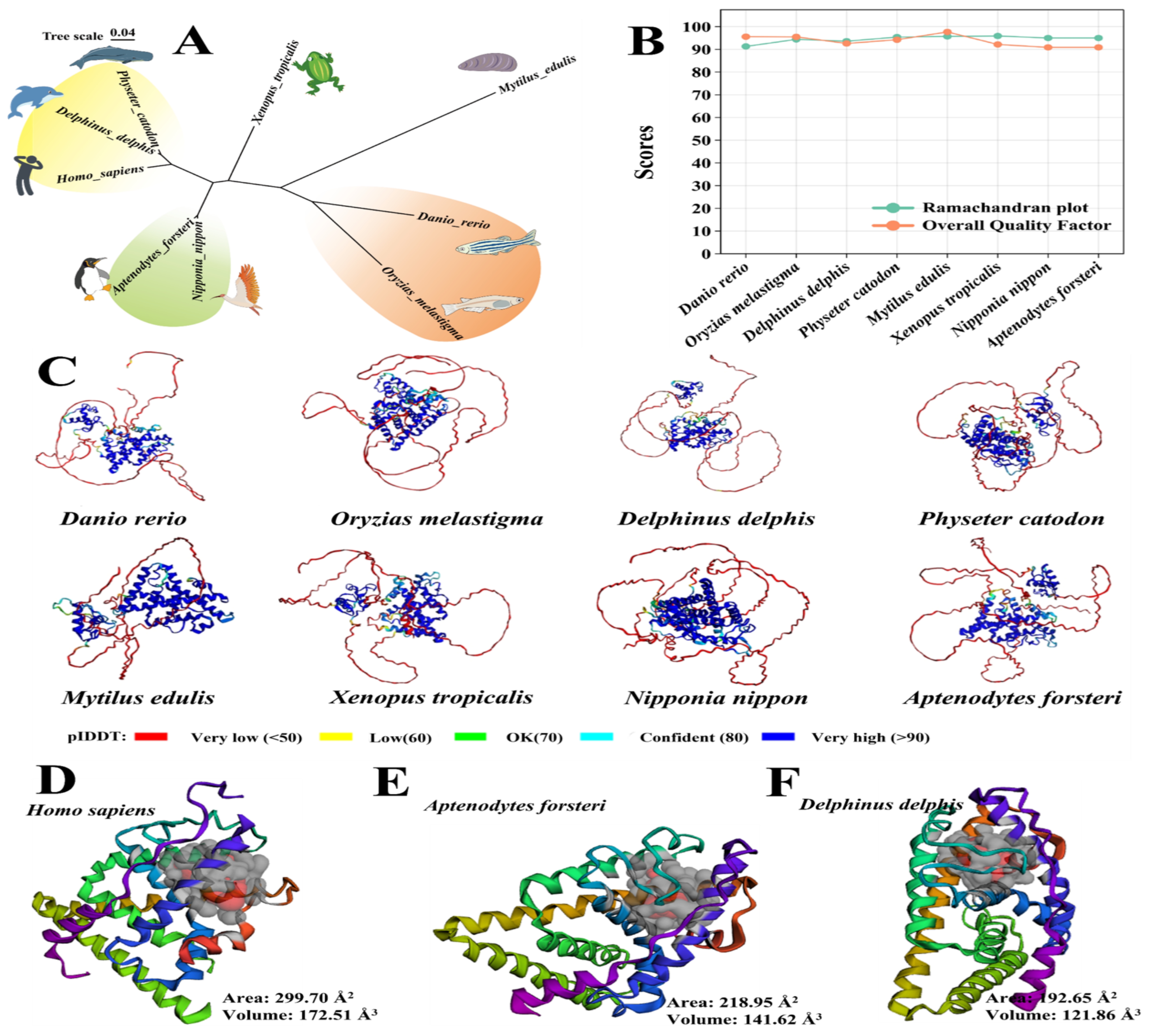
图3. 不同物种ERα序列进化树分析与3D结构构建。(A) 进化树分析。(B) 不同物种的ERα三维结构的质量评估。(C) 不同物种的ERα三维结构示意图。
ERα的三维结构:如图3C所示,预测的物种ERα LBD的pLDDT分数均表现出较高的置信水平。Ramachandran图中Dr、Om、Dd、Pc、Me、Xt、Nn和Af的残基位于优选区域的百分比分别为91.3%、94.4%、93.6%、95.4%、95.7%、95.9%、95%和95%。这些模型的整体质量因子分别为95.57、95.49、92.60、94.24、97.66、92.17、90.86和90.86(图3B)。
6. ERα激动剂结合的关键氨基酸
为了突出不同物种间关键氨基酸及其保守性,展示的是根据LBD序列进行比对后氨基酸的编号(SI_Text 表 S10)。ER全长氨基酸序列号与LBD中序列号的对应关系见SI_Text 表 S10。简而言之,谷氨酸 (Glu) 44 和精氨酸 (Arg) 85 (对应人类ER全长序列中的Glu353和Arg394)是大多数物种中负责ERα活性的两个关键氨基酸残基(图4A,SI_Text 表 S10)。在与水生相关的物种中,还鉴定出另外三个残基:甘氨酸 (Gly) 212,亮氨酸 (Leu) 216 和苯丙氨酸 (Phe) 95(图4A,SI_Text 表 S10)。包括 Glu44/43/41、Arg85/84/82 和 Gly212/211/197 在内的这些关键残基在不同物种间高度保守(图4B)。典型的ERα激动剂(包括BPTMC和雌激素)与各物种ERα之间的相互作用见SI_Dataset 表 S9。除Me外,BPTMC和雌激素主要与所有物种ERα中的Arg85或Glu44形成氢键。在Me中,BPTMC和雌激素与ERα中的色氨酸 (Trp) 201 形成氢键。有趣的是,筛选出的具有ERα激动剂活性的塑料添加剂主要与 Arg85/84、Glu44/43/41 和 Phe95/92 形成氢键(图4C,SI_Text 表 S11)。在Me中,Trp 201为ERα活性的关键残基(图4A,4C)。
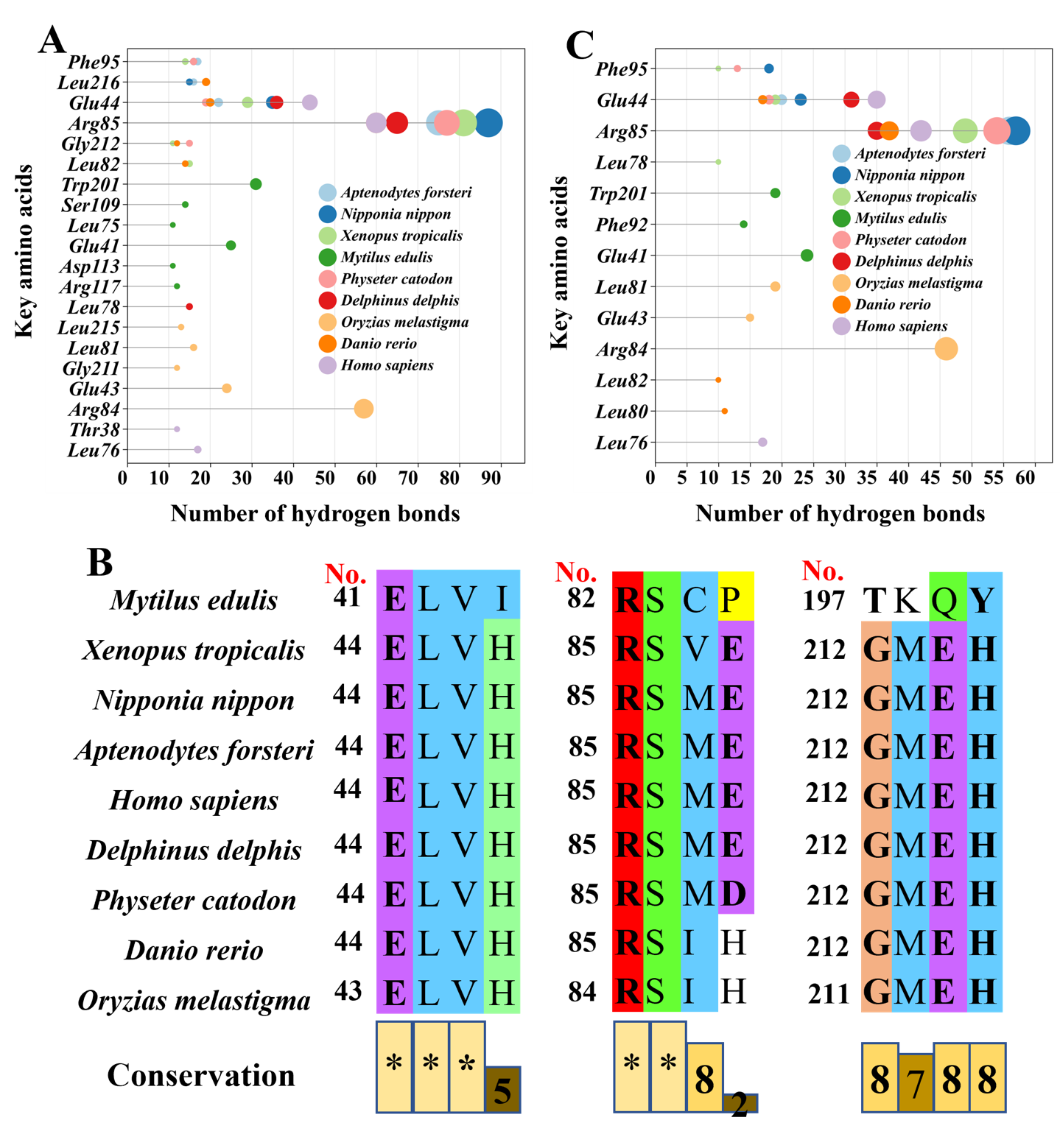
图4. 识别与ERα激动剂相互作用的关键氨基酸。(A) 每个物种ERα中的关键氨基酸。为研究与典型ERα激动剂相互作用的氨基酸,通过分子对接将训练数据集中所有化合物与不同物种的ERα进行分析,量化了与ERα激动剂形成氢键的氨基酸的频率。(B) 不同物种关键氨基酸的保守性,其中谷氨酸、精氨酸和甘氨酸显示高度保守。(C) 所筛选的塑料添加剂与9个物种ERα中形成氢键的关键氨基酸。为了方便,图中的氨基酸编号均是基于LBD中氨基酸序列比对后的编号,突出显示了关键氨基酸及其跨物种的保守性(SI_Text 表S10)。
7. 可疑ER激动剂与各物种ERα的结合能:结合能的聚类分析显示,8种脊椎动物和1种无脊椎动物形成了两条独立的分支。在脊椎动物中,人类与海洋动物聚为一组,而鱼类和水鸟形成了另一组(图5A)。这些发现与进化树分析的结果相一致。筛选出的塑料添加剂与ERα相互作用的平均结合能分别为:Hs(−7.84 kcal/mol)、Dr(−7.31 kcal/mol)、Om(−7.58 kcal/mol)、Dd(−7.49 kcal/mol)、Pc(−7.85 kcal/mol)、Me(−7.67 kcal/mol)、Xt(−7.94 kcal/mol)、Nn(−7.65 kcal/mol)和Af(−7.72 kcal/mol)(见SI_Text表S12)。
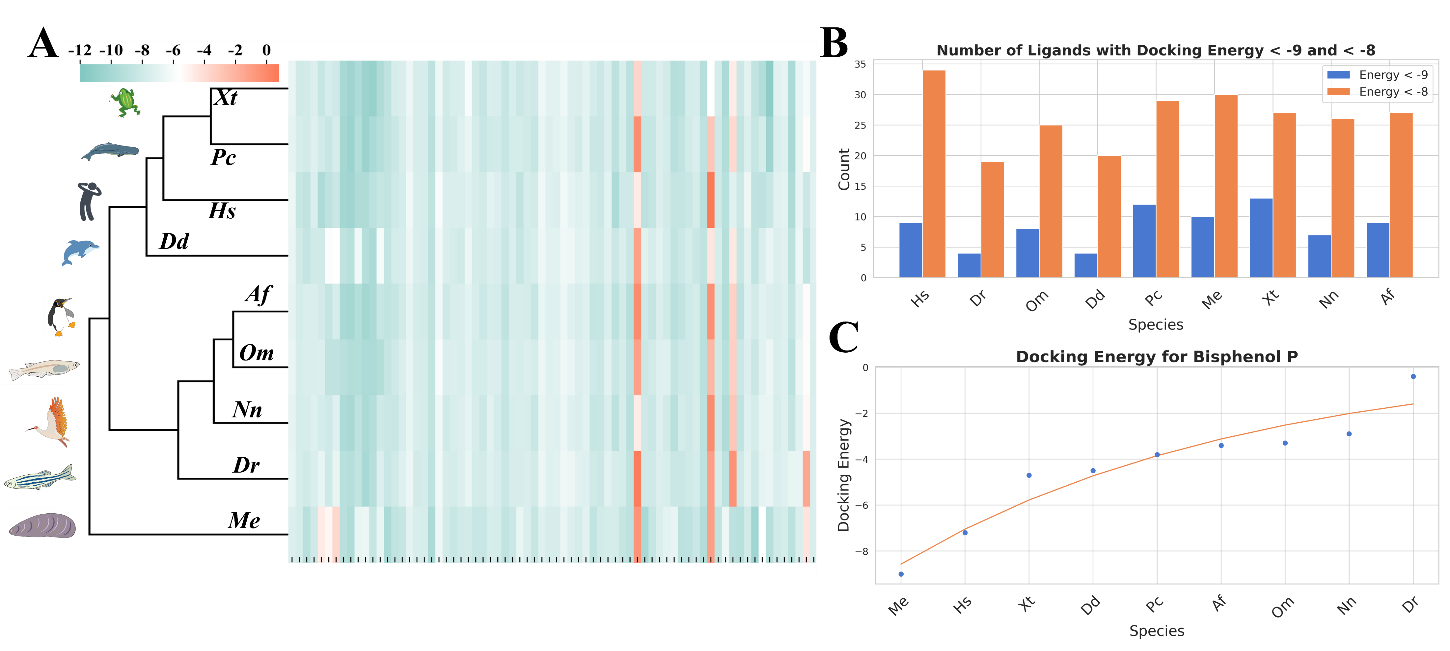
图5. (A) 不同物种结合能的聚类分析。(B) 不同物种在不同结合能阈值下的化合物数量。(C) 双酚P在不同物种中的结合能。Hs: 人类;Dr: 斑马鱼;Om: 青鳉鱼;Dd: 海豚;Pc: 抹香鲸;Me: 贻贝;Xt: 非洲爪蟾;Nn: 朱鹮;Af: 帝企鹅。
在不同物种Hs、Dr、Om、Dd、Pc、Me、Xt、Nn和Af的ERα中,共有9、4、8、4、12、10、13、7和9个塑料添加剂与这些物种ERα分子对接能量低于−9 kcal/mol(图5B,附表S10)。此外,分别有34、19、25、20、29、30、27、26和27个塑料添加剂与这些物种ERα分子对接能量低于−8 kcal/mol(图5B)。雌酮在所有物种中均表现出低于−9 kcal/mol的对接能量,而双酚P在不同物种之间的对接能量呈现显著差异(图5C)。另外,多种塑料添加剂,包括2,2-双(4-羟基-3-甲基苯基)丙烷、4-(2-萘氨基)苯酚、1,1-双(4-羟基苯基)环己烷、双酚AF、以及4,5-双酚-o-醌,其与所有物种ER的对接能量均低于−8 kcal/mol。
结论与意义
在本研究中,我们使用机器学习方法筛选ERα激动剂,并利用AlphaFold2构建了8种不同物种的ERα结构。确定了与大多数物种雌激素受体结合的两个关键氨基酸残基(Arg85和Glu44,对应人类ER序列中Arg394和Glu353)对这些物种的ERα活性具有重要的影响。在脊椎动物和无脊椎动物之间,观察到分子对接能量的显著差异。水生相关物种正日益暴露于各种污染物中,对其健康及周边生态系统构成重大威胁。评估新污染物对多种物种的毒性对于理解其对生态系统的影响至关重要。目前,体内实验仍然是评估毒性的主要方法,但这些方法成本高且耗时长,限制了实验效率。为解决这些挑战,利用人工智能技术预测污染物在不同物种中的毒性成为一种备受关注的替代方案。在本研究中,结合AlphaFold2和机器学习技术为高通量预测新污染物与不同物种受体蛋白的亲和力提供了新的策略。
全文信息
Wen-Jun Shi, Zhou Cao, Xiao-Bing Long, Chong-Rui Yao, Jin-Ge Zhang, Chang-Er Chen, Guang-Guo Ying. Predicting estrogen receptor agonists from plastic additives across various aquatic-related species using machine learning and AlphaFold2. Journal of Hazardous Materials 2025, 494, 138629.
Xiao-Bing Long, Chong-Rui Yao, Si-Ying Li, Jin-Ge Zhang, Zhi-Jie Lu, Dong-Dong Ma, Chang-Er Chen, Guang-Guo Ying, Wen-Jun Shi*. Screening androgen receptor agonists of fish species using machine learning and molecular model in NORMAN water-relevant list. Journal of Hazardous Materials 2024, 468, 133844.